이진 분류
이진 분류(Binary Classification) :
임의의 샘플 데이터를 참 혹은 거짓으로 구분하는 문제를 말한다.
퍼셉트론(Perceptron) :
이전에 공부한 선형 회귀와 매우 유사함 하지만 퍼셉트론은 마지막 단계에서 샘플을 이진 분류하기 위해 계단 함수를 사용한다.
이후 계단 함수를 통과한 값을 다시 가중치와 절편을 갱신하도록 학습하는데 사용한다.
만약 뉴런은 입력 신호들을 받아 z를 만든다고 해보자
$w_1x_1 + w_2x_2 + b = z$라 하자
아니 이전에 공부한 선형 방정식과 다른 것 같은데 -> 이전에는 wx + b = y였는데 이 것은 입력값이 한개 더 늘었을 뿐이다. (원래 x 한개에서 $x_1과x_2$ 2개의 featrue가 입력값)
자 이제 선형 함수의 출력값인 z를 계단함수에 적용해보자.. 그런데 계단함수가 뭐지?
계단 함수(step function) :
z가 0보다 크거나 같으면 1로 0보다 작으면 -1로 분류하는 함수
y = 1 (z >= 0)
y = 0 (z < 0)
image_reference : aistudy.co.kr/neural/activation_function.htm
정리 하자면 퍼셉트론은 선형 함수를 통과해서 얻은 결과값인 z를 계단함수에 입력하여 그 값이 0보다 클지 작을지에 대해 검사하여 크다면 1을 작다면 -1로 분류하는 간단한 알고리즘 이다.
퍼셉트론은 계단 함수의 결과를 사용하여 가중치와 절편을 업데이트 한다.
현재부터 여러 개의 특성(feature)을 사용해 보자
그러면 특성이 n개인 선형 함수를 나타내보면
$y=w_1x_1=w_2x_2+~~~+w_nx_n+b$
이 식을 sigma기호로 나타내면
$b + \sum_{i=1}^{n} w_ix_i$
늘어난 입력 특성에 따라 가중치(w) 갯수도 따라 늘어나는군… 당연하지 입력(x)에 곱해지는 값이 가중치인데 물론 절편(b)은 1개 그대로임
지금까지 퍼셉트론에 대해 알아 보았다.
지금까지 퍼셉트론을 모두 구현해 왔지만 사이킷런 패키지에서 Perceptron이라는 이름의 클래스를 제공해 주기 때문에 이 것을 이용하면 직접 구현할 필요가 없다.
그럼 지금까지 뭐한거 ?? 어떻게 해당 클래스가 구성되어있는지 알면 그 클래스를 사용하거나 이해하기 좋으니까..
아달린(Adaline) :
퍼셉트론을 개선한 적응형 선형 뉴런(Adaptive Linear Neuron)
적응형 선형 뉴런 ??? 그냥 선형 뉴런과 무슨 차이 ??
아달린에 대해 알아보면 아달린은 선형 함수의 결과를 학습하는데 사용한다 (선형 함수의 가중치와 절편 갱신하는 학습). 그리고 계단 함수의 결과는 예측에만 활용한다….
이전에 배운 선형 뉴런 경우 선형 함수의 결과를 계단 함수에 입력하고 그 결과를 이용해 학습과 예측을 하였다. 하지만 아달린 경우에는 역방향 게산이 계단 함수 출력 이후가 아닌 선형 함수의 출력 이후에 진행된다는 차이점이 있다.
그런데 아달린을 하면 뭐가 더 좋아지나 보군.. 어떤것이??
아달린을 좀더 개선한 버전이 로지스틱 회귀이다.
로지스틱 회귀가 이전 선형 뉴런에 비해 어떤 점이 좋은지 알 수 있으면 위의 의문이 해결될 것이다.
로지스틱 회귀(logistic regression) :
아달린에서 좀더 발전한 형태이다.
로지스틱 회귀는 선형 함수를 통과시켜 얻은 z를 임계 함수에 보내기 전에 변형시키는데 이 변형 시켜주는 함수가 활성화 함수(activation function)이다.
선형 함수를 통과하여 얻은 z가 활성화 함수에 들어가여 그 결과로 a를 내놓는다고 하자 그리고 이 a는 임계 함수에 들어가 예측값을 얻게 된다.
또한 아달린과 유사하게 (좀 다름) a가 역방향 계산이 일어나 선형함수의 결과를 학습하는데 사용된다 (적합한 가중치와 절편 찾는 학습)
임계 함수는 아달린에서의 계단 함수와 역할 비슷하지만 활성화 함수의 출력값을 사용한다는 점이 차이다.
각 차이점을 정리 해보자
퍼셉트론 (일반 선형 뉴런) :
입력 -> 선형 함수 -> z -> 계단 함수 -> y_hat
예측 값인 y_hat가 다시 역방향 계산이 일어나 선형 함수의 결과를 학습하는데 사용 (적합한 가중치와 절편 찾는 학습)
아달린 (적응형 선형 뉴런) :
입력 값 -> 선형 함수 -> z -> 계단 함수 -> y_hat
선형 함수의 결과값인 z가 다시 역방향 계산이 일어나 선형 함수의 결과를 학습하는데 사용 (적합한 가중치와 절편 찾는 학습)
로지스틱 회귀 :
입력값 -> 선형 함수 -> z -> 활성화 함수 -> a -> 게단 함수 -> y_hat
활성화 함수의 결과값인 a가 다시 역방향 계산이 일어나 선형 함수의 결과를 학습하는데 사용 (적합한 가중치와 절편 찾는 학습)
활성화 함수는 비선형 함수를 사용해야 한다.!!
왜 활성화 함수로는 선형함수가 아닌 비선형 함수를 사용해야 하지 ??
ex) 만약 선형함수인 $y = w_1x_1 + w_2x_2 + …. + w_nx_n$에다가 활성화 함수 y = ka (선형 함수) 가 있다고 하자.
이 둘을 쌓은 수식은 $y = k(w_1x_1 + w_2x_2 + … + w_nx_n)$이 되고 이 결과는 다시 선형함수가 된다. 이렇게 되면 임계 함수 앞에 뉴런을 아무리 많이 쌓는다고 해도 그 결과가 선형함수일 것이고 그로 인해 큰 의미가 생기지 않게된다. 따라서 활성화 함수로 비선형 함수를 사용하는 것이다.
비선형 함수의 예로 $p = \frac{1}{1 + e^{-z}}$ 이 있으며 이 외에도 다양한 함수가 존재한다.
그러면 로지스틱 회귀에서 사용되는 활성화 함수는 어떤 함수일까?
-> 로지스특 회귀에서는 “시그모이드 함수(sigmoid function)”를 활성화 함수로 사용한다.
시그모이드 함수가 어떤 역할을 하며, 어떤 과정으로 만들어지겓 되었는지 알아보자
로지스틱 회귀에서 선형함수의 출력값 z는 $z = b + \sum_{i=1}^{n} w_ix_i$인데 이 z 값을 활성화 함수를 통과시켜서 a가 되게 된다. 이때 시그모이드 함수는 z를 0 ~ 1 사이의 확률값으로 변환시켜주는 역할을 하게 한다.
예를들어 분류를 해야하는 경우에 시그모이드 함수의 결과인 a가 0.5(50%)보다 크면 양의 클래스, 그 이하가 되면 음의 클래스로 분류를 한다.
그럼 시그모이드 함수가 어떻게 만들어 지는지 과정에 대해 알아보자
오즈 비 -> 로짓 함수 -> 시그모이드 함수
오즈 비(odds ratio) :
성공 확률과 실패 확률의 비율을 나타내는 통계
OR(odds ratio) = $\frac{p}{1 - p}$ (p = 성공 확률)
오즈피를 그래프로 나타내면
image_reference : https://kau-deeperent.tistory.com/90
로짓 함수(logit function) :
오즈 비에 로그함수를 취하여 만든 함수
logit(p) = $log(\frac{p}{1 - p})$
로짓 함수는 p = 0.5일때 0이 되고 p가 0과 1일때 각각 무한대로 음수와 양수가 되는 특징을 가진 함수이다.
그래프로 나타내보면
image_reference : https://i-am-eden.tistory.com/21
로짓 함수의 세로 축을 z로 가로축을 p로 놓으면 확률 p가 0 에서 1까지 변할 때 z가 매우 큰 음수에서 매우 큰 양수로 증가하는 것을 볼 수 있다.
따라서 이 식을 다시 정리하면
z = log($\frac{p}{1 - p}$)
로지스틱 함수(Logistic function) :
위 로직 함수 z에 대해 아래와 같이 정리하면 로지스틱 함수가 된다.
$p = \frac{1}{1 + e^{-z}}$
이 식을 유도한 방법은 기존 로짓함수의 식에서 z = ~ 꼴이 아닌 p = ~꼴로 바꾸어 주는 것이다.
이렇게 정리한 이유는 기존 로잣함수의 가로 축이 p 였는데 가로 축을 z로 놓기 위해서 이다. (역함수 변환과 유사)
image_reference
이렇게 나타낸 로지스틱 함수를 그래프로 그려보자
로짓 함수의 가로와 세로축을 반대로 뒤집은 모양이 된다. -> 로짓 함수의 역함수가 로지스틱 함수 (y = x 그래프 기준 대칭으로 보면 됨.)
그리고 이 그래프는 S자 형태를 띈다.
이 S자 형태를 착안해서 로지스틱 함수를 시그모이드 함수(Sigmoid Function) 이라고 부른다.
로지스틱 회귀를 정리 해보면
선형 함수 -> z -> 로지스틱 함수 -> a -> 임계 함수 -> y_hat
a는 역방향 계산으로 선형함수의 가중치와 절편을 갱신함.
이때 z는 $-\infty$ ~ $\infty$의 범위를 가지는데 로지스틱 회귀는 이진 분류가 목적이기 때문에 z 범위를 조절해야 한다. 이 문제의 해결을 위해 시그모이드 함수를 활성화 함수로 사용하여 0 ~ 1사이의 값으로 범위를 조절하였다.
따라서 시그모이드 함수의 결과인 a값이 0 ~ 1 로 확률처럼 해석이 가능하게 되었다. a를 확률로 이해하면 a를 0과 1로 구분 (이진 분류) 하기 위해서 마지막에 임계 함수를 이용하였다.
임계 함수에서 a값이 0.5보다 큰지 혹은 이하인지에 따라 1 혹은 0으로 y_hat으로 결과를 내놓았다.
지금 까지 이진 분류를 어떻게 하는지는 알겠는데 그럼 a 값으로 역방향 계산을 통해 선형함수의 가중치와 절편을 어떻게 갱신하는지 궁금하다…
이 방법도 손실함수를 사용할 것이다. 그러면 로지스틱 회귀에서는 어떤 손실 함수를 사용해 가중치와 절편을 갱신할 수 있을까??
이전에 배운 선형회귀에서 손실 함수로 제곱 오차를 사용한 것처럼 로지스틱 회기에서도 적용 안되려나 ??
이전에 배운 선형회귀는 정답과 예상값의 오차 제곱이 최소가 되느 가중치와 절편을 찾는 방법을 이용하였다 (제곱 오차) 하지만 로지스틱 회귀에서는 선형 회귀와 목적이 다르다. 선형 회귀는 어떤 값을 찾는 거라면 로지스틱 회귀는 분류가 목적이다!!!
즉 올바르게 분류를 하는 비율을 높이는 것이 목표이다.
이전에 배운 방법인 경사 하강법의 손실함수를 사용하려고 하니 … 올바르게 분류된 샘플의 비율은 미분 가능한 함수가 아니기 때문에 이 방법은 불가능 하다. 그러면 다른 함수를 사용해야 한다..
-> 이 함수가 바로 “로지스틱 손실 함수” 이다.
로지스틱 손실 함수 :
다중 분류를 위한 손실 함수인 크로스 엔트로피 (Cross Entropy) 손실 함수를 이진 분류 버전으로 만든 것
크로스 엔트로피 손실 함수는 이후에 배울 것이다.
L = -(ylog(a) + (1 - y)log(1 - a))
로지스틱 회귀는 이진분류에가 목적이기 때문에 y값 (타깃값)이 0 혹은 1이다. 따라서 위 손실함수 식에서 y값이 0 혹은 1이 되다.
y가 0인 경우 (음성 클래스) -> L = -log(1 - a)
y가 1인 경우 (양성 클래스) -> L = -log(a)
위 두 식의 값을 최소로 만들다 보면 a의 값이 원하는 목표의 값에 가까워 진다는 것을 알 수 있다.
음성 클래스 경우 (y = 0 경우) 로지스틱 손실 함수의 값(L)을 최소로 만들려면 a는 0에 가까워 지게 되고
양성 클래스 경우 (y = 1 경우) 로지스틱 손실 함수의 값(L)을 최소로 만들려면 a는 1에 가까워 지기 때문이다.
이 값들 (다시 을 계단 함수에 통과시키면 올바르게 분류 작업이 수행 하게 된다.
정리 하자면 로지스틱 손실 함수를 최소화 하면 a 값이 우리가 원하는 값이 되게 된다.
로지스틱 손실 함수의 미분
로지스틱 회귀의 가중치와 절편을 갱신하하기 위해 로지스틱 손실 함수를 미분해보자
미분 하는 이유는 나중에 알 수 있다.
가중치와 절편에 대한 로지스틱 손실 함수의 미분 결과는
$\frac{\delta}{\delta w_i}L = -(y-a)x_i$
$\frac{\delta}{\delta b }L = -(y-a)x_i$
이전 선형회귀 에서의 가중치와 절편에 대한 미분 결과와 유사하다
다만 y_hat 대신에 a가 있을 뿐이다.
로지스틱 회귀의 구현이 사실 선형 회귀와 큰 차이가 없는 것 같다.
우리는 로지스틱 손실함수의 가중치에 대한 미분, 절편에 대한 미분을 해야하는데
이 미분을 어떻게 해야할지 고민이다. 왜냐하면 로지스틱 손실함수(L)을 가중치(w)나 절편(b)로 바로 미분하는 것은 너무 복잡하기 때문이다.
미분의 연쇄 법칙을 이용한다면 이 문제를 해결할 수 있다.
미분의 연쇄법칙 :
$\frac{\delta y}{\delta x} =\frac{\delta y}{\delta u} \frac{\delta u}{\delta x}$
미분의 연쇄 법칙을 적용하기 이전에 로지스틱 회귀의 과정을 다시 정리해보자
그 이유로는 역방향 계산 방향을 알아야 미분의 연쇄법칙을 적용할 수 있기 때문이다.
입력($x_1, x_2, … x_n$) -> 선형 함수($w_1, w_2, —, w_n$, b) -> z -> 활성화 함수 -> a -> 계단함수 -> y_hat
a에서 역방향 계산을 하여 선형 함수의 가중치와 절편을 갱신하고 그렇게 되면 더욱 타깃에 적합한 z->a->y_hat 값을 찾게 될 것이다.
이 과정을 보면 로지스틱 손실 함수에 대한 미분이 연쇄 법칙에 의해 진행되는 구조이다.
이러한 구조를 </b> Gradient가 역전되었다 </b>라고 표현한다.
그럼 이제 다시 본론으로 돌아와 미분의 연쇄법칙을 적용해 로지스틱 손실함수의 가중치, 절편에 대한 미분을 해보자
먼저 로지스틱 손실함수(L)을 활성화 함수의 출력값(a)에 대해 미분하고
활성화 함수의 출력값(a)은 선형 함수의 출력값(z)에 대하여 미분하고
선형 함수의 출력값(z)은 가중치(w) 또는 절편(b)에 대해 미분한다.
이 미분한 결과들을 서로 곱해주면 원하는 로지스틱 손실함수의 가중치 혹은 절편에 대해 미분한 결과가 된다.
이 과정이 미분의 연쇄법칙을 이용한 것이다.
이제 다시 정리해보면
로지스틱 손실 함수를 가중치에 대한 미분 :
$\frac{\delta}{\delta w_i }L = \frac{\delta L}{\delta a }\frac{\delta a}{\delta z }\frac{\delta z}{\delta w_i }$
로지스틱 손실 함수를 절편에 대한 미분 :
$\frac{\delta}{\delta b}L = \frac{\delta L}{\delta a }\frac{\delta a}{\delta z }\frac{\delta z}{\delta b }$
위 두 미분 계산 과정을 생략하고 식을 정리하면
$\frac{\delta}{\delta w_i }L = -(y - a)x_i$
$\frac{\delta}{\delta b}L = -(y - a)1$
이다.
이제 미분을 완료 하였으니 가중치와 절편을 갱신해보도록 하자.
로지스틱 회귀의 가중치 갱신
로지스틱 회귀의 가중치를 갱신하기 위해 로지스틱 손실 함수를 가중치에 대해 미분한 식을 가중치에서 뺀다.
$w_i = w_i - \frac{\delta L}{\delta w_i} = w_i + (y-a)x_i$
로지스틱 회귀의 절편 갱신
가중치 갱시과 유사한 방법으로
$b = b - \frac{\delta L}{\delta b} = (y-a)1$
이전에 가중치, 절편 갱신을 위해 왜 로지스틱 손실함수에 가중치와 절편에 대해 미분해야 했는지 이유를 위의 갱신하는 방법에서 쓰이기 때문인것을 알 수 있다. -> 로지스틱 손실함수에 대한 가중치와 절편의 변화율을 이용
이전 선형 회귀에서 경사하강법을 기억할 것이다. 그때 왜 가중치(w), 절편(b)의 변화율을 이용하여 절편과 가중치를 갱신하는 이유를 설명하였다.
위에서 설명한 이진 분류를 이젠 직접 데이터를 이용해 구현해볼 것이다.
유방암 데이터를 이용할 것인데 이 유방암 데이터에는 특징이 10개가 존재한다.
평균, 표준오차, 최대 이상치 등이 잇는데 이를 이용해 유방암 데이터 샘플이 악성 종양(True)인지 정상 종양(False) 인지 판단해 볼 것이다. (이진 분류)
# 유방암 데이터 세트를 준비한다.
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
print(cancer)
# cancer를 보면 처음에 입력 데이터, 두번째 0, 1로 타겟 데이터, 세번쨰는 뭐지??
{'data': array([[1.799e+01, 1.038e+01, 1.228e+02, ..., 2.654e-01, 4.601e-01,
1.189e-01],
[2.057e+01, 1.777e+01, 1.329e+02, ..., 1.860e-01, 2.750e-01,
8.902e-02],
[1.969e+01, 2.125e+01, 1.300e+02, ..., 2.430e-01, 3.613e-01,
8.758e-02],
...,
[1.660e+01, 2.808e+01, 1.083e+02, ..., 1.418e-01, 2.218e-01,
7.820e-02],
[2.060e+01, 2.933e+01, 1.401e+02, ..., 2.650e-01, 4.087e-01,
1.240e-01],
[7.760e+00, 2.454e+01, 4.792e+01, ..., 0.000e+00, 2.871e-01,
7.039e-02]]), 'target': array([0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0,
0, 0, 1, 0, 1, 1, 1, 1, 1, 0, 0, 1, 0, 0, 1, 1, 1, 1, 0, 1, 0, 0,
1, 1, 1, 1, 0, 1, 0, 0, 1, 0, 1, 0, 0, 1, 1, 1, 0, 0, 1, 0, 0, 0,
1, 1, 1, 0, 1, 1, 0, 0, 1, 1, 1, 0, 0, 1, 1, 1, 1, 0, 1, 1, 0, 1,
1, 1, 1, 1, 1, 1, 1, 0, 0, 0, 1, 0, 0, 1, 1, 1, 0, 0, 1, 0, 1, 0,
0, 1, 0, 0, 1, 1, 0, 1, 1, 0, 1, 1, 1, 1, 0, 1, 1, 1, 1, 1, 1, 1,
1, 1, 0, 1, 1, 1, 1, 0, 0, 1, 0, 1, 1, 0, 0, 1, 1, 0, 0, 1, 1, 1,
1, 0, 1, 1, 0, 0, 0, 1, 0, 1, 0, 1, 1, 1, 0, 1, 1, 0, 0, 1, 0, 0,
0, 0, 1, 0, 0, 0, 1, 0, 1, 0, 1, 1, 0, 1, 0, 0, 0, 0, 1, 1, 0, 0,
1, 1, 1, 0, 1, 1, 1, 1, 1, 0, 0, 1, 1, 0, 1, 1, 0, 0, 1, 0, 1, 1,
1, 1, 0, 1, 1, 1, 1, 1, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 1, 1, 1, 1, 1, 1, 0, 1, 0, 1, 1, 0, 1, 1, 0, 1, 0, 0, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 1, 1, 0, 1, 0, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 1, 1, 1, 0, 1, 0, 1, 1, 1, 1, 0, 0,
0, 1, 1, 1, 1, 0, 1, 0, 1, 0, 1, 1, 1, 0, 1, 1, 1, 1, 1, 1, 1, 0,
0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 0, 1, 0, 0, 0, 1, 0, 0,
1, 1, 1, 1, 1, 0, 1, 1, 1, 1, 1, 0, 1, 1, 1, 0, 1, 1, 0, 0, 1, 1,
1, 1, 1, 1, 0, 1, 1, 1, 1, 1, 1, 1, 0, 1, 1, 1, 1, 1, 0, 1, 1, 0,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 1, 0, 0, 1, 0, 1, 1, 1, 1,
1, 0, 1, 1, 0, 1, 0, 1, 1, 0, 1, 0, 1, 1, 1, 1, 1, 1, 1, 1, 0, 0,
1, 1, 1, 1, 1, 1, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 1, 1, 1, 1,
1, 1, 1, 0, 1, 0, 1, 1, 0, 1, 1, 1, 1, 1, 0, 0, 1, 0, 1, 0, 1, 1,
1, 1, 1, 0, 1, 1, 0, 1, 0, 1, 0, 0, 1, 1, 1, 0, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 0, 1, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 0, 0, 0, 0, 0, 1]), 'target_names': array(['malignant', 'benign'], dtype='<U9'), 'DESCR': '.. _breast_cancer_dataset:\n\nBreast cancer wisconsin (diagnostic) dataset\n--------------------------------------------\n\n**Data Set Characteristics:**\n\n :Number of Instances: 569\n\n :Number of Attributes: 30 numeric, predictive attributes and the class\n\n :Attribute Information:\n - radius (mean of distances from center to points on the perimeter)\n - texture (standard deviation of gray-scale values)\n - perimeter\n - area\n - smoothness (local variation in radius lengths)\n - compactness (perimeter^2 / area - 1.0)\n - concavity (severity of concave portions of the contour)\n - concave points (number of concave portions of the contour)\n - symmetry \n - fractal dimension ("coastline approximation" - 1)\n\n The mean, standard error, and "worst" or largest (mean of the three\n largest values) of these features were computed for each image,\n resulting in 30 features. For instance, field 3 is Mean Radius, field\n 13 is Radius SE, field 23 is Worst Radius.\n\n - class:\n - WDBC-Malignant\n - WDBC-Benign\n\n :Summary Statistics:\n\n ===================================== ====== ======\n Min Max\n ===================================== ====== ======\n radius (mean): 6.981 28.11\n texture (mean): 9.71 39.28\n perimeter (mean): 43.79 188.5\n area (mean): 143.5 2501.0\n smoothness (mean): 0.053 0.163\n compactness (mean): 0.019 0.345\n concavity (mean): 0.0 0.427\n concave points (mean): 0.0 0.201\n symmetry (mean): 0.106 0.304\n fractal dimension (mean): 0.05 0.097\n radius (standard error): 0.112 2.873\n texture (standard error): 0.36 4.885\n perimeter (standard error): 0.757 21.98\n area (standard error): 6.802 542.2\n smoothness (standard error): 0.002 0.031\n compactness (standard error): 0.002 0.135\n concavity (standard error): 0.0 0.396\n concave points (standard error): 0.0 0.053\n symmetry (standard error): 0.008 0.079\n fractal dimension (standard error): 0.001 0.03\n radius (worst): 7.93 36.04\n texture (worst): 12.02 49.54\n perimeter (worst): 50.41 251.2\n area (worst): 185.2 4254.0\n smoothness (worst): 0.071 0.223\n compactness (worst): 0.027 1.058\n concavity (worst): 0.0 1.252\n concave points (worst): 0.0 0.291\n symmetry (worst): 0.156 0.664\n fractal dimension (worst): 0.055 0.208\n ===================================== ====== ======\n\n :Missing Attribute Values: None\n\n :Class Distribution: 212 - Malignant, 357 - Benign\n\n :Creator: Dr. William H. Wolberg, W. Nick Street, Olvi L. Mangasarian\n\n :Donor: Nick Street\n\n :Date: November, 1995\n\nThis is a copy of UCI ML Breast Cancer Wisconsin (Diagnostic) datasets.\nhttps://goo.gl/U2Uwz2\n\nFeatures are computed from a digitized image of a fine needle\naspirate (FNA) of a breast mass. They describe\ncharacteristics of the cell nuclei present in the image.\n\nSeparating plane described above was obtained using\nMultisurface Method-Tree (MSM-T) [K. P. Bennett, "Decision Tree\nConstruction Via Linear Programming." Proceedings of the 4th\nMidwest Artificial Intelligence and Cognitive Science Society,\npp. 97-101, 1992], a classification method which uses linear\nprogramming to construct a decision tree. Relevant features\nwere selected using an exhaustive search in the space of 1-4\nfeatures and 1-3 separating planes.\n\nThe actual linear program used to obtain the separating plane\nin the 3-dimensional space is that described in:\n[K. P. Bennett and O. L. Mangasarian: "Robust Linear\nProgramming Discrimination of Two Linearly Inseparable Sets",\nOptimization Methods and Software 1, 1992, 23-34].\n\nThis database is also available through the UW CS ftp server:\n\nftp ftp.cs.wisc.edu\ncd math-prog/cpo-dataset/machine-learn/WDBC/\n\n.. topic:: References\n\n - W.N. Street, W.H. Wolberg and O.L. Mangasarian. Nuclear feature extraction \n for breast tumor diagnosis. IS&T/SPIE 1993 International Symposium on \n Electronic Imaging: Science and Technology, volume 1905, pages 861-870,\n San Jose, CA, 1993.\n - O.L. Mangasarian, W.N. Street and W.H. Wolberg. Breast cancer diagnosis and \n prognosis via linear programming. Operations Research, 43(4), pages 570-577, \n July-August 1995.\n - W.H. Wolberg, W.N. Street, and O.L. Mangasarian. Machine learning techniques\n to diagnose breast cancer from fine-needle aspirates. Cancer Letters 77 (1994) \n 163-171.', 'feature_names': array(['mean radius', 'mean texture', 'mean perimeter', 'mean area',
'mean smoothness', 'mean compactness', 'mean concavity',
'mean concave points', 'mean symmetry', 'mean fractal dimension',
'radius error', 'texture error', 'perimeter error', 'area error',
'smoothness error', 'compactness error', 'concavity error',
'concave points error', 'symmetry error',
'fractal dimension error', 'worst radius', 'worst texture',
'worst perimeter', 'worst area', 'worst smoothness',
'worst compactness', 'worst concavity', 'worst concave points',
'worst symmetry', 'worst fractal dimension'], dtype='<U23'), 'filename': '/usr/local/lib/python3.7/dist-packages/sklearn/datasets/data/breast_cancer.csv'}
# 입력 데이터를 확인해 보자.
print(cancer.data.shape, cancer.target.shape) #입력 데이터의 data 크기를 출력한다.
#569개 샘플있고 30개의 특성이 있음을 확인할 수 있다.
(569, 30) (569,)
cancer.data[:3] #샘플 3개만 봐보자
#특성들이 실수 범위에 양수값이네 정도만 알 수 있다.
#이전 선형 회귀 처럼 산점도로 나타내서 특성간의 관계에 대해 보고 싶은데... 이게 30개나 되니까 산점도로 표현하기는 힘들듯..
array([[1.799e+01, 1.038e+01, 1.228e+02, 1.001e+03, 1.184e-01, 2.776e-01,
3.001e-01, 1.471e-01, 2.419e-01, 7.871e-02, 1.095e+00, 9.053e-01,
8.589e+00, 1.534e+02, 6.399e-03, 4.904e-02, 5.373e-02, 1.587e-02,
3.003e-02, 6.193e-03, 2.538e+01, 1.733e+01, 1.846e+02, 2.019e+03,
1.622e-01, 6.656e-01, 7.119e-01, 2.654e-01, 4.601e-01, 1.189e-01],
[2.057e+01, 1.777e+01, 1.329e+02, 1.326e+03, 8.474e-02, 7.864e-02,
8.690e-02, 7.017e-02, 1.812e-01, 5.667e-02, 5.435e-01, 7.339e-01,
3.398e+00, 7.408e+01, 5.225e-03, 1.308e-02, 1.860e-02, 1.340e-02,
1.389e-02, 3.532e-03, 2.499e+01, 2.341e+01, 1.588e+02, 1.956e+03,
1.238e-01, 1.866e-01, 2.416e-01, 1.860e-01, 2.750e-01, 8.902e-02],
[1.969e+01, 2.125e+01, 1.300e+02, 1.203e+03, 1.096e-01, 1.599e-01,
1.974e-01, 1.279e-01, 2.069e-01, 5.999e-02, 7.456e-01, 7.869e-01,
4.585e+00, 9.403e+01, 6.150e-03, 4.006e-02, 3.832e-02, 2.058e-02,
2.250e-02, 4.571e-03, 2.357e+01, 2.553e+01, 1.525e+02, 1.709e+03,
1.444e-01, 4.245e-01, 4.504e-01, 2.430e-01, 3.613e-01, 8.758e-02]])
import matplotlib.pyplot as plt
plt.boxplot(cancer.data)
plt.xlabel('feature')
plt.ylabel('value')
plt.show()
# 산점도로 나타내기 힘드니 박스플롯으로 나타내 보았다. (박스플롯은 나중에 설명)
# 박스 플롯을 보면 4, 14, 24번째 특성에 대한 value값이 다른 특성들보다 분포가 훨씬 넓게 퍼저있음을 알 수 있다.
# -> 왜 이런지 한번 알아보자
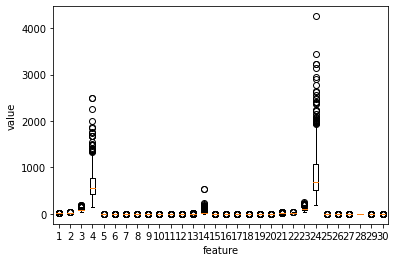
cancer.feature_names[[3, 13, 23]]
# 4, 14, 24번째 (인덱스는 0부터니 코드랑 햇갈리지 말길)의 특성을 보면
# area : 넓이와 관련된 것임을 알 수 있다.
# 아 ... 주어진 데이터에서 세포들의 넓이가 다양하나 보다..
array(['mean area', 'area error', 'worst area'], dtype='<U23')
박스 플롯 :
image_reference : http://demo.riamore.net/HTML5demo/chart/Docs/User%20Manual%20-%20html/box-plot-chart.html
위 그림에서 하위 표본 퀀타일이 1사분위, 표본 미디언이 2사분위, 사위 표본 퀀타일이 3사분위로 보면 된다.
박스 플롯은 1사분위와 3사분위 값으로 상자를 그리고 그 안에 2사분위값을 나타낸다.
1사분위와 3사분위 사이의 거리의 1.5배만큼 위아래 거리를 그려놓은 것이다.
import numpy as np
np.unique(cancer.target, return_counts = True) #타깃 데이터 확인
# 0과 1로만 우리어져 있고 0은 음성클래스 1은 양성클래스를 나타냄
# numpy의 unique()함수는 고유한 값을 찾아 반환하는 것이고
# 이때 return_counts = True로 매개변수 지정을 하면 고유한 값이 얼만큼 있는지 횟수를 반환 한다.
# -> 결과를 통해 음성클래스(정상 종양) 212개, 양성클래스(악성 종양) 357개가 있음을 알 수 있다.
print(357 / 212)
# 양성클래스 : 음성클래스 비율이 대충 1.7 : 1 로 확인된다. -> 이 비율 기억해두길 !!!
1.6839622641509433
# 훈련 데이터 세트 저장
x = cancer.data
y = cancer.target
이전에 선형 회귀로 뉴런 클래스 만든 것 기억해보면 훈련 데이터 세트를 주어진 데이터 전체를 이용하여 모델을 훈련했다.
사실 웃긴 일이다. 모든 데이터로 훈련을 하면 어떤 데이터로 모델이 잘 훈련 되었는지 알 수 있을까?
훈련 데이터로 다시 훈련이 잘 되었는지 검증하는 것은 ??
예전에 읽은 책 중에 “혼자 공부하는 머신러닝, 딥러닝”에서 한 예시가 기억이 난다.
어떤 사람에게 특정 과목에 대해 잘 알 수 있게 훈련 문제들로 훈련을 시키고 과연 잘 공부 되었는지 확인하기위해 시험을 보았는데 시험 문제로 이전의 훈련 문제들로 시험문제를 낸다면 ?
해당 과목의 전체적인 공부가 잘 되지 않았더라도 훈련 문제만을 기억해서 풀어 모두 맞출수도 있기 떄문에 훈련이 잘 되었는지 제대로 확인할 수 없다.
이 예시와 마찬가지로 훈련 데이터로 검증을 한다?? 정확한 검증을 할 수 없다.
그러면 훈련 데이터의 모든 데이터를 모델을 훈련 시키기 위한 훈련 세트와 모델이 잘 훈련이 되었는지 검증할 테스트 세트로 나누면 될 것이다.
이렇게 나눈 것을
훈련 세트(training set)
테스트 세트(test set)
라고 한다.
그런데 어떻게 나눌까?? 그냥 주어진 데이터 반으로 딱 나누면 안되나?
-> 아니다 훈련 데이터 세트를 훈련 세트와 테스트 세트로 나누는 2가지 규칙이 존재한다.
훈련 데이터 세트를 훈련 세트와 테스트 세트로 나누는 규칙
- 훈련 데이터 세트를 나눌 때는 테스트 세트보다 훈련 세트가 많아야 함
- 훈련 데이터 세트를 나누기 전에 양성 클래스와 음성 클래스가 훈련 세트나 테스트 세트 한쪽에 몰려선 안된다. (편향이 있어선 안된다.)
이러한 규칙을 지키지 않ㅇ면 데이터에 있는 패턴을 제대로 학습하지 못하기 때문에 잘못된 측정을 할 수 있다.
하… 이런 규칙을 지키면서 데이터 나누기 너무 귀찮은데..
-> 사이킷런의 도구를 이용하면 편ㄴ하다.
사이킷런을 이용해 훈련 데이터 세트를 훈련 세트와 테스트 세트로 나누어 보자.
# 사이킷런을 이용해 훈련 데이터 세트를 훈련세트와 테스트 세트로 나누어 보자
from sklearn.model_selection import train_test_split
# sklearn.model_selection 모듈에 있는 train_test_split() 함수는 기본적으로 입력된 훈련 데이터 세트를 훈련 세트를 75%, 테스트 세트를 25% 비율로 나눈다.
x_train, x_test, y_train, y_test = train_test_split(x, y, stratify = y, test_size = 0.2, random_state = 42)
# 매개 변수에 대해서 알아보면
# 1. stratify = y :
# stratify는 훈련 데이터를 나눌 때 클래스 비율을 동일하게 하는 매개변수이다.
# train_test_split은 default로 데이터를 나누기 전에 섞지만 일부 클래스 비율이 균형이 맞지 않은 경우에 stratifyfmf y로 지정해야 한다.
# 2. test_size = 0.2 :
# train_test_split()함수는 default로 훈련 데이터 세트를 75 : 25 비율로 나눈다.
# 하지만 test_size 매개변수를 이용하면 이 비율을 조절할 수 있다.
# 이 예제처럼 test_size = 0.2로 하면 훈련 데이터 세트를 80 : 20 비율로 나눌 수 있다.
# 3. random_state = 42 :
# train_test_split() 함수는 무작위로 데이터 세트를 섞은 다음 나누는데 (랜덤하게),
# 이때 random_state는 rando seed를 세팅한다.
# 랜덤 값은 사실 엄격한 랜덤값이 아니다.
# 어떤 특정한 시작 숫자를 정해 주면 컴퓨터가 정해진 알고리즘에 의해 마치 난수처럼 보이는 수열을 생성한다.
# 이런 시작 숫자를 시드(seed)라고 한다.
# 예제와 같이 random_state = 42로 난수 초기값을 지정해 두면 데이터 세트를 섞는 결과가 항상 일정하게 나타난다.
# 훈련 데이터 세트가 잘 나누어졌는지 훈련 세트와 테스트 세트의 비율을 확인해 보자
print(x_train.shape, x_test.shape)
print(455 / 114)
# 결과를 확인해 보니 훈련 세트와 테스트 세트 80 : 20 비율로 잘 나누어져 있는것을 확인할 수 있다.
(455, 30) (114, 30)
3.991228070175439
# numpy의 unique()함수를 이용해서 훈련 세트의 타깃 안에 있는 클래스의 갯수를 확인해 보자.
np.unique(y_train, return_counts = True)
print(285 / 170)
# 확인해보니 전체 훈련 데이터 세트의 클래스 비율과 거의 비슷한 것을 알 수 있다.
# -> 양성클래스 : 음성클래스 비율이 약 1.7 : 1 정도 됨
# 이전에 훈련 데이터를 나누기전에 양성클래스 : 음성클래스 비율을 기억해두라 한 적이 있다. 그 이유가 그때도 비율이 약 1.7 : 1
# 정도 였는데 train_test_split()함수가 훈련 데이터 세트를 훈련 세트와 테스트 세트로 나누는 규칙인 훈련 중
# 훈련 데이터 세트를 나누기 전에 양성, 음성 클래스가 훈련 세트나 테슽 ㅡ세트의 어느 한쪽에 몰리지 않도록 골고루 섞어야 한다는 규칙을
# 잘 지킨 것으로 확인된다.
1.6764705882352942
# 지금까지 배운 로지스틱 회귀를 이 데이터로 구현해보자
class LogisticNeuron:
def __init__(self):
self.w = None
self.b = None
# -> 생성자에서 특이한점은 가중치와 절편을 미리 초기화 하지 않는다는 점이다.
# 그 이유는 입력 데이터의 특성 갯수를 알지 못하므로
# 가중치를 입력데이터가 들어오고 난 후 특성 개수에 맞게 결정
def forpass(self, x): # 선형 방정식 계산 (로지스틱 회귀에서 데이터가 정방향으로 흘러가는 과정)
z = np.sum(x * self.w) + self.b
return z
def backprop(self, x, err): # 역방향 계산 (가중치와 절편을 업데이트 하기 위해 데이터가 역방향으로 흘러가는 과정)
w_grad = x * err # 가중치에 대한 gradient 계산
b_grad = 1 * err # 절편에 대한 gradeint 계산
return w_grad, b_grad
# 훈련하는 메서드를 구현해보자
# 훈련 수행하는 fit() 메서드를 만들 것인데 이전 선형 회귀에서 구현한 Neuron 클래스에서 만든 함수와 같다
# 하지만 차이점으로는 activation()메서드가 추가되었음 -> 당연 선형회귀와 로지스틱회귀의 차이점을 생각하면 알 수 있음
def fit(self, x, y, epochs = 100):
self.w = np.ones(x.shape[1]) #가중치를 초기화 한다. np.ones()함수를 이용해 1로 초기화
self.b = 0 #절편을 0으로 초기화
for i in range (epochs): # epochs만큼 반복
for x_i, y_i in zip(x, y):
z = self.forpass(x_i) # 선형함수식에 대입하여 z 구함
a = self.activation(z) # z를 활성화 함수에 대입하여 a 구함
err = -(y_i - a) # 오차를 계산함
w_grad, b_grad = self.backprop(x_i, err) # 역방향 계산으로 가중치 gradient와 절편 gradient 구함
self.w -= w_grad # 가중치를 갱신한다.
self.b -= b_grad # 절편을 갱신한다.
# activation() 메서드를 만들어보자
# 로지스틱 회귀에서 사용할 활성화 함수는 시그모이드 함수이다. 따라서 시그모이드 함수를 구현하면 된다.
def activation(self, z):
a = 1 / (1 + np.exp(-z))
return a
# 예측값을 구해보자 (y_hat 구하기)
# 이전 선형 회귀 Neuron 클래스 구현에서 새로운 샘플에 대한 예측값 구할때 forpass() 메서드 사용했던것 기억할 것이다.
# 근데 만약 에측해 볼 샘플이 많다면 forpass()메서드를 계속 호출해야하는 귀찮은 일이 생긴다.
# 거기다가 로지스틱 회귀에서는 활성화 함수와 임계 함수까지 추가되니 어휴... 정말 귀찮다.
# -> 이러한 문제를 해결해줄 predict()메서드를 만들어 보자
# 샘플에 대한 예측값을 계산해주는 predict() 메서드를 만들어 보자
def predict(self, x):
z = [self.forpass(x_i) for x_i in x] # 리스트 컴프리헨션으로 반복문으로 여러 샘플을 선형 함수에 대입 후 그 결과를 리스트에 저장
a = self.activation(np.array(z)) # 활성화 함수에 z 리스트 값 대입
return a > 0.5 # 계단 함수를 적용 함!!!!
# predict()메서드의 매개변수 값으로 입력값 x가 2차원 배열로 전달 되었다고 가정하겠음
# -> 매개변수 x에 이전에 만들어둔 x_train이 들어올 것인데
# x_train.shape = (455, 30) 즉 샘플 455개인데 각 샘플당 특성이 30개인 데이터 이므로
# 2차원 배열로 이루어 져 있을 것이다.[[샘플1], [샘플 2], [샘플 3], [] ~~~ ] (각 샘플당 30개의 특성)
# ex) 샘플 1 = [1, 2, ~~~ , 30] (꼭 숫자가 1, 2, 3 ~~ 이 아니라 갯수를 나타낸 것임)
# 로지스틱 회귀 모델을 훈련시켜 보자
# 모델을 훈련해보자
neuron = LogisticNeuron() # LogisticNeuron에 대한 객체를 만듬 (객체를 모델이라고 부르기도 함)
neuron.fit(x_train, y_train) # 이전에 만들어둔 훈련 세트로 (입력, 타깃 훈련 세트) 모델 훈련시키기
/usr/local/lib/python3.7/dist-packages/ipykernel_launcher.py:49: RuntimeWarning: overflow encountered in exp
# 테스트 세트 사용해서 모델의 정확도를 평가해 보자
np.mean(neuron.predict(x_test) == y_test)
# predict() 메서드의 반환값은 Ture 혹은 False가 원소인 (m, ) 크기의 배열
# y_test는 0 또는 1이 원소인 (m, ) 크기의 배열이기 때문에 비교 가능
# np.mean() 함수 : 매개변수 값으로 전달한 비교문 결과의 평균값을 계산하여 반환 -> 이것을 정확도(accuracy)라고 한다.
/usr/local/lib/python3.7/dist-packages/ipykernel_launcher.py:49: RuntimeWarning: overflow encountered in exp
0.8245614035087719
지금까지 로지스틱 회귀에 대해 직접 구현해 보았다.
사실 지금 구현한 로지스틱 회귀 뉴런이 단일층 신경망이다.
구체적인 내용(층에 대한 내용 등등)은 이후에 설명해 볼 것이다.
또한 로지스틱 회귀를 구현하는데 생각보다 귀찮은데 이 부분 또한 사이킷런에서 제공하는 클래스를 이용하면 편리하다.
이 부분도 이후에 설명할 것이다.
Reference
박해선, 딥러닝 입문, 이지스퍼블리싱, 2019, 76~104pg